Sukces polskich naukowców w modelowaniu białek
Nauka w Polsce (PAP) | 2005-01-07
 Trzech polskich naukowców znalazło się w gronie zwycięzców 6. edycji światowego, prestiżowego konkursu w modelowaniu struktury przestrzennej białek, odbywającego się w Stanach Zjednoczonych pod nazwą Critical Assesment of Techniques for Protein Structure Prediction (CASP).
Trzech polskich naukowców znalazło się w gronie zwycięzców 6. edycji światowego, prestiżowego konkursu w modelowaniu struktury przestrzennej białek, odbywającego się w Stanach Zjednoczonych pod nazwą Critical Assesment of Techniques for Protein Structure Prediction (CASP).
Wyróżnieni naukowcy to: prof. Andrzej Koliński z Wydziału Chemii UW, dr Krzysztof Ginalski z Interdyscyplinarnego Centrum Modelowania Matematycznego i Komputerowego UW i BioInfoBank Institute oraz dr Janusz Bujnicki z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej.
Zbadanie przestrzennej struktury jak największej liczby białek ma nie tylko znaczenie czysto poznawcze. Otwiera też nieograniczone pole zastosowań praktycznych, wśród których najważniejsze wydaje się projektowanie leków - mówi dr Krzysztof Ginalski, który po raz drugi został nieoficjalnym liderem tegorocznej edycji konkursu CASP.
Poznanie trójwymiarowej budowy białek, tzn. sposobu zwinięcia się nici białka do pewnego unikatowego kształtu w przestrzeni, jest absolutnie niezbędne do poznania roli, jaką to białko spełnia w organizmie. "Tylko ten określony kształt wykazuje działanie biologicznie poprawne" - podkreśla prof. Lucjan Piela z Wydziału Chemii UW.
Do tej pory naukowcom udało się doświadczalnie poznać budowę przestrzenną około 20 tys. białek i określić ponad 2 mln sekwencji białkowych.
Badanie struktury białek metodami doświadczalnymi jest jednak bardzo czasochłonne i drogie. Określenie sposobu przestrzennego ułożenia się sekwencji aminokwasów może potrwać niekiedy nawet kilka lat.
Pomocne okazują się tu metody teoretyczne, tzw. modelowanie komputerowe. Wykorzystywane do tego celu programy pozwalają znacznie szybciej - często w ciągu kilku godzin - na zaproponowanie sposobu zwinięcia się nici wybranych sekwencji aminokwasów.
Zadaniem uczestników konkursu jest zaproponowanie za pomocą symulacji komputerowej struktur przestrzennych dla podanych sekwencji aminokwasów.
Źródło: Nauka w Polsce
------
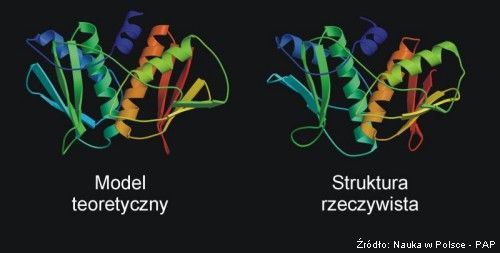
Na schemacie:
Najdokładniejszy model struktury jednego z białek (wystawiony do konkursu CASP6, opracowany przez dr Krzysztofa Ginalskiego) oraz jego prawdziwy odpowiednik.
Źródło schematu: Nauka w Polsce (PAP)
Komentarze
Cerebra | 2005-02-10 00:00:00
Polak potrafi!! Super chlopaki!! Tak trzymac!!! Dzieki Wam wierze w polskich naukowców!!!!! (bo na rzad nie ma co liczyc- banda idiotów) Chociaz Wy jestescie duma narodu polskiego!!!
Ja tez tak chce jak Wy!! Choc droga dluga i kreta, bede sie starac!!!
