Pirosekwencjonowanie - sekwencjonowanie z wykorzystaniem kwantów światła
Karolina Chwiałkowska | 2009-02-20
W przeciągu ostatnich dekad nastąpił ogromny rozwój biologii molekularnej. Stało się jasnym, że nie jest możliwe zbadanie wszystkich interakcji zachodzących w komórkach, bez poznania ich molekularnego podłoża zawartego między innymi w sekwencji DNA poszczególnych genów. Pierwsza ważna sekwencja DNA została odczytana w 1971 roku przez Wu i Taylora. Był to kohezyjny koniec DNA faga λ. W 1977 roku zostały opracowane niezależnie dwie metody wyznaczania sekwencji DNA. Pierwsza z nich - metoda Sangera - to metoda wykorzystująca terminację łańcucha DNA przy zastosowaniu zmodyfikowanych nukleotydów (dideoksynukleotydy). Druga metoda - Maxima i Gilberta - polega na wykorzystaniu związków chemicznych do rozszczepiania łańcucha DNA. Obie techniki są powszechnie stosowane w wielu laboratoriach po dziś dzień. W 1996 roku w Royal Institute of Technology w Sztokholmie profesor Pal Nyren, wraz ze swoim uczniem Mostafa Ronaghi, opracował nowatorską technikę sekwencjonowania DNA - pirosekwencjonowanie.
Pirosekwencjonowanie to metoda sekwencjonowania DNA w czasie rzeczywistym. W reakcji pirosekwencjonowania biorą udział cztery enzymy, którymi są: fragment Klenowa polimerazy DNA I, sulfurylaza ATP, lucyferaza i apiraza. Reakcje katalizowane przez te enzymy są związane z sobą – produkty kolejnych są substratami następnych. Matryca to jednoniciowe fragmenty DNA. Cząsteczki te są zazwyczaj produktami łańcuchowej reakcji polimerazy (PCR), która ma na celu namnożenie wybranych fragmentów DNA. Primery jednej z nici muszą być wyznakowane biotyną, co pozwala na separację jednoniciowych odcinków dzięki związaniu ich ze streptawidyną, która ma silne powinowactwo do biotyny. Do separacji jednoniciowych odcinków, wykorzystuje się specjalne urządzenie zasysające oraz końcówki z filtrem, na którym osadza się złoże sefarozowe opłaszczone streptawidyną. Badane odcinki DNA wiążą się z tym złożem, a kolejne etapy denaturacji i odpłukania powodują, iż w efekcie uzyskuje się jednoniciowe matryce. Są one następnie uwalniane ze złoża do specjalnej płytki z roztworem starterów sekwencyjnych. Związania starterów z matrycami dokonuje się poprzez ogrzanie płytki w 90 st. C, a następnie schłodzeniu jej do 45 st. C. Tak przygotowaną płytkę umieszcza się w urządzeniu sekwencjonującym, uzupełnia dozowniki reagentami i uruchamia sekwentator. Od tej pory dalsze etapy wykonywane są przez maszynę automatycznie.
Pierwszą zachodzącą reakcją jest polimeryzacja nici DNA zachodząca po dodaniu jednego z czterech nukleotydów. Jeśli nukleotyd jest komplementarny do matrycy DNA, to zostaje on wbudowany w nowopowstającą nić. Niekomplementarne nukleotydy zostają zdegradowane przez inny enzym. Reakcję polimeryzacji, przedstawioną poniżej, katalizuje fragment Klenowa polimerazy DNA I.
Fragment Klenowa zawiera oddzielone od siebie przestrzennie aktywności polimerazy i egzonukleazy 3’->5’, lecz nie zawiera aktywności egzonukleazowej 5’->3’. W reakcji pirosekwencjonowania, obecność aktywności egzonukleazowej jest nie tylko niepotrzebna, lecz również może ona mieć negatywny wpływ na jej przebieg poprzez wycinanie pewnych odcinków DNA. Możliwe jest również, dzięki mutacji powodującej zmianę dwóch aminokwasów, zlikwidowanie aktywności egzonukleazowej 3’->5’ z fragmentu Klenowa bez naruszania jego struktury. W reakcjach pirosekwencjonowania, w celu uniknięcia niesynchronicznej polimeryzacji DNA, używa się właśnie takich zmodyfikowanych fragmentów Klenowa. Dzięki rozpoznawaniu przez enzym przestrzennego ułożenia struktury DNA determinowanego komplementarnością par zasad, brak tej aktywności nie zwiększa znacznie błędów podczas polimeryzacji.
Kolejną z reakcji w technologii pirosekwencjonowania jest produkcja ATP z PPi uwalnianego podczas polimeryzacji nici DNA. Reakcja ta jest katalizowana przez sulfurylazę ATP. Enzym ten może być otrzymywany z bakterii Aquifex pyrophilius. Sulfurylaza ATP przeprowadza reakcję w obecności atomów magnezu. Kataliza obejmuje reakcję (przedstawioną na schemacie poniżej) pomiędzy pirofosforanem a adenozynofosfosiarczanem (APS).
Reszta kwasu siarkowego (VI) z cząsteczki APS zostaje zastąpiona pirofosforanem. Powstaje wolny kwas siarkowy (VI) oraz adenozynotrifosforan (ATP) niezbędny w dalszych reakcjach.
Nieprzereagowany ATP oraz nukleotydy (dNTP) ulegają reakcji odłączania reszt fosforanowych katalizowanej przez apirazę. Tri- i difosforany nukleotydów zostają przekształcane w monofosforany. Przykład reakcji z ATP jako substratem przedstawiono poniżej.
Kolejne reszty fosforanowe są odszczepiane, lecz tylko te, które są związane
z następną z nich. Ostatnia, przyłączona do rybozy, nie jest odłączana, co skutkuje powstaniem monofosforanu adenozyny (AMP). Reakcji tej ulega zarówno nadmiar ATP, jak i niewykorzystane nukleotydy. Wykorzystanie apirazy powoduje zmniejszenie tła reakcji, gdyż powstałe monofosforany nie są rozpoznawane przez żaden z enzymów i mogą współistnieć w mieszaninie nie zakłócając przebiegu żadnego z procesów.
Powstały, w reakcji przeprowadzanej przez sulfurylazę, ATP ulega konwersji w kolejnych dwóch reakcjach katalizowanych przez lucyferazę. W pierwszej z nich bierze udział w przekształceniu lucyferyny w lucyferylo-AMP:
W następnej reakcji związana lucyferyna ulega utlenieniu. Powstaje oksylucyferyna w stanie wzbudzonym, która przy przejściu w stan podstawowy emituje strumień fotonów (hv).
Reakcja pirosekwencjonowania, ze względu na niską termostabilność lucyferazy, powinna być przeprowadzana w temperaturze poniżej 25 st. C. Jednakże w wyższej temperaturze występuje optimum działania pozostałych enzymów stosowanych w sekwencjonowaniu. Skróceniu ulega czas reakcji oraz zmniejszają się sygnały tła reakcji. Stąd też ważne jest podjęcie działań mających na celu zwiększenie termostabilności lucyferaz. Dowiedziono, że lucyferaza, z podstawioną leucyną w pozycji 217 zamiast treoniny, wykazuje zwiększoną termostabilność. Dzieje się tak dlatego, że zamiana hydrofilowego aminokwasu, jakim jest treonina, na hydrofobową leucynę indukuje zmiany przestrzenne w cząsteczce.
Reakcji aktywacji lucyferyny, z wytworzeniem lucyferyloadenozynomonofosforanu związanego z enzymem, ulega zarówno D-lucyferyna jak i L-lucyferyna. Lucyferaza posiada zdolność produkcji światła jedynie z deoksyadenozynotrifosforanu (dATP). Aby poprawić tło reakcji stosuje się α-thio-dATP jako substrat dla polimerazy, która posiada zdolność katalizy reakcji z wykorzystaniem, w przeciwieństwie do lucyferazy, tego nukleotydu. Zapobiega to wystąpieniu sygnału świetlnego po dodaniu do mieszaniny reakcyjnej dATP.
Reakcji utlenienia lucyferyny (będącej związaną z AMP i lucyferazą) tlenem cząsteczkowym z wydzieleniem cząsteczki dwutlenku węgla oraz AMP reakcji z wydzieleniem kwantów światła ulega jedynie D-lucyferyna. Reakcja z L-lucyferyną jako substratem jest reakcją „ciemną”.
Reakcjom przedstawionym powyżej ulega 80% lucyferyny, pozostałe 20% poddaje się w drugim etapie reakcji ubocznej przedstawionej poniżej (pierwszy etap to związanie enzymu z substratem i dołączenie AMP). Lucyferyloadenozynomonofosforan związany z enzymem(E◦LH2-AMP) ulega reakcji z tlenem cząsteczkowym tworząc dehydrolucyferyloadenozynomonofosforan(L-AMP) z wydzieleniem cząsteczki nadtlenku wodoru.
W reakcji przeprowadzanej przez lucyferazę w temperaturze 25 st. C, po dodaniu do roztworu wszystkich niezbędnych składników, występuje początkowo faza spoczynkowa (lag). Dopiero po 25 ms następuje emisja pierwszych fotonów, która stopniowo wzrasta, osiągając maksimum w 2 s reakcji, a następnie spada, osiągając połowę maksymalnej emisji w dziesiątej sekundzie. W czasie fazy lag zachodzi związanie enzymu z substratem i reakcja pomiędzy ATP a lucyferyną z wytworzeniem LH2-AMP. Spadek emisji światła jest wywołany gromadzącymi się produktami (oksylucyferyna, L-AMP), które są inhibitorami lucyferazy. Wykazano, że populacja wolnego inhibitora maleje w trakcie tworzenia się kompleksów z enzymem, co sugeruje, że L-AMP może nieodwracalnie wiązać enzym, powodując zmiany konformacyjne uniemożliwiające związanie substratu i zajście dalszych reakcji.
Sekwentator automatycznie dozuje reagenty do mieszaniny, a wyniki wyświetlane są w czasie rzeczywistym na ekranie komputera. Szybkość reakcji to jedna minuta na jeden wbudowany nukleotyd. Poszczególne próbki w dołkach płytki analizowane są w tym samym czasie niezależnie od siebie. Emitowany sygnał świetlny zostaje wychwycony przez zamontowaną kamerę, a informacja przesłana do podłączonego komputera. Intensywność piku świetlnego zostaje przełożona, przy pomocy odpowiedniego oprogramowania, na sekwencję nukleotydową badanego odcinka DNA.
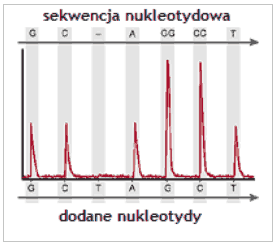
Rys.1. Zależność intensywności piku świetlnego od ilości wbudowanych nukleotydów
Źródło: http://www.biotagebio.com/DynPage.aspx?id=7454 (strona czynna w dniu 15.02.2009.) - zmienione
Jak obrazuje przedstawiony powyżej wykres, im wyższy jest pik świetlny, tym większa ilość nukleotydów została wbudowana kolejno po sobie. Brak sygnału świetlnego świadczy o tym, że nukleotyd nie został wbudowanym z powodu niekomplementarności. Po zakończeniu sekwencjonowania, wynik jest zapisywany w pliku, który można poddać dalszej analizie.
Pirosekwencjonowanie cechuje wiele zalet, którymi są:
Czułość - analiza występujących mutacji punktowych obejmujących zmianę tylko jednego nukleotydu.
Specyficzność - dzięki zastosowaniu reakcji PCR oraz znakowania biotyną starterów, próbka podlegająca sekwencjonowaniu zawiera tylko i wyłącznie badany fragment DNA.
Redukcja kosztów - koszt zsekwencjoncjonowania pojedynczej próbki przy zastosowaniu technologii pirosekwencjonowania wynosi 69 centów, natomiast przy jednoczesnym sekwencjonowaniu dużej ilości prób, spada aż do 20 centów.
Automatyzacja - dzięki wykorzystaniu podłączonej do sekwentatora kamery i specjalnego oprogramowania, pozwala na niemalże całkowitą automatyzację procesu. Wymagane jest jedynie odpowiednie przygotowanie próbki do sekwencjonowania oraz uzupełnienie dozowników reagentami. Cała procedura sekwencjonowania wykonywana jest przez urządzenie.
Szybkość analizy - przy wykorzystaniu pirosekwencjonowania, czas upływający od pobrania próbki, który obejmuje izolację materiału genetycznego, jego namnożenie oraz przygotowanie, wynosi kilka godzin. Zastosowanie standardowego sekwencjonowania, wydłuża ten proces do paru dni.
Ilościowa korelacja - intensywność sygnału podczas pirosekwencjonowania jest determinowana ilością występujących po sobie kolejno tych samych nukleotydów.
Pirosekwencjonowanie znalazło zastosowanie w takich analizach jak: badanie sekwencji odcinków DNA, wykrywanie mutacji i polimorfizmów pojedynczych nukleotydów, genotypowanie wirusów i bakterii, badanie mitochondrialnego DNA oraz wielu innych.
Literatura:
1. A. Ahmadian et al.: Clin Chim Acta. 363: 83, 2006.
2. M. DeLuca, W. McElroy: Biochemistry. 13: 921, 1974.
3. B. Gharizadeh et al.: Anal Biochem. 301: 82, 2002.
4. E. Hanna et al.: Arch Biochem Biophys. 406: 275, 2002.
5. T. Hopkins et al.: J Am Chem Soc. 91: 2178, 1969.
6. N. Kajiyama, E. Nakano: Biochemistry. 32: 13795, 1993.
7. H. Klenow, K. Overgaard-Hansen: FEBS Lett. 6: 25, 1970.
8. M. Komoszyński, A. Wojtczak: Biochim Biophys Acta. 1310: 233, 1996.
9. N. Pourmand et al.: Nucl Acids Res. 30: e31, 2002.
10. C. Ribeiro, J. Esteves da Silva: Photochem Photobiol Sci. 7: 1085, 2008.
11. M. Ronaghi: Genome Res. 11: 3, 2001.
12. R. Słomski et al.: Przykłady Analiz DNA. Wydawnictwo Akademii Rolniczej im. Augusta Cieszkowskiego w Poznaniu, Poznań: 2004.
13. http://www.biotagebio.com/DynPage.aspx?id=7454 (strona czynna w dniu 15.02.2009.)
Autor: Karolina Chwiałkowska
Kontakt: karolina.chwialkowska(at)gmail.com
